Efectos de un programa de entrenamiento aeróbico en el tiempo de reacción y la metilación de ADN del gen SHANK3 en niños con trastorno de espectro autista: un estudio piloto
Effects of an Aerobic Training Program on Reaction Time and DNA Methylation of SHANK3 Gene in Children with Autism Spectrum Disorder: a Pilot Study
Érika Yolanda Hernández Sandoval , Karim-Martina Alvis-Gómez, Humberto Arboleda Granados, Ana-Gabriela Concha-Mera
Resumen
Introducción: El trastorno del espectro autista (TEA) es un trastorno del neurodesarrollo que provoca déficits en áreas cognitivas y motoras y es causado por varios mecanismos, entre ellos la regulación epigenética. Los procesos epigenéticos pueden verse influenciados por factores ambientales como el ejercicio físico.
Objetivo: Analizar el efecto de un programa de ejercicio físico aeróbico (EFA) en el tiempo de reacción simple (TRS) y la metilación del ADN de la isla 2 del gen SHANK3 en niños con TEA.
Materiales y métodos: Estudio cuasiexperimental realizado con un grupo de 9 niños (7-11 años) con TEA, que participaron en un programa de EFA de 10 semanas. Las diferencias en el TRS y la metilación de ADN fueron analizadas mediante la prueba de Kruskall-Wallis, considerando un nivel de significancia de p‹0.05.
Resultados: La mediana del TRS disminuyó después del programa de entrenamiento. Sin embargo, no se encontró una diferencia estadísticamente significativa (p=0.53). Se observó un patrón de hipermetilación en 11 de los dinucleótidos, tanto antes como después del entrenamiento, y se encontró una diferencia estadísticamente significativa en la posición CpG108 (p=0.032).
Conclusión: Un programa de entrenamiento basado en EFA de intensidad moderada a vigorosa tiene el potencial de modificar el TRS y la metilación del ADN en niños con TEA. No obstante, es necesario realizar nuevos estudios con muestras más grandes y en los que se analicen más genes, para corroborar los resultados aquí descritos y fortalecer el conocimiento sobre el efecto del ejercicio en los procesos epigenéticos de esta población.
Palabras clave
Trastorno del espectro autista; metilación de ADN; entrenamiento aeróbico; ejercicio físico; tiempo de reacción.
Abstract
Introduction: Autism spectrum disorder (ASD) is a neurodevelopmental disorder that produces cognitive and motor deficits and it is caused by several mechanisms, including epigenetic regulation. Epigenetic processes can be influenced by environmental factors such as physical exercise.
Objective: To analyze the effect of an aerobic physical exercise (APE) program on simple reaction time (SRT) and DNA methylation of island 2 of the SHANK3 gene in children with ASD.
Materials and methods: A quasi-experimental study was carried out on a group of 9 children (7-11 years old) with ASD, who participated in a 10-week APE program. Differences in SRT and DNA methylation were analyzed using the Kruskall-Wallis test by considering a significance level p‹0.05.
Results: The median SRT decreased after the training program. However, no statistically significant difference was found (p = 0.53). A pattern of hypermethylation was observed in 11 dinucleotides, both before and after training, and a statistically significant difference was found in the CpG108 position (p = 0.032).
Conclusion: A moderate to vigorous intensity of APE program has the potential to modify SRT and DNA methylation in children with ASD. However, it requires further studies with larger samples in which more genes are analyzed, to corroborate the results described here and strengthen knowledge about the effect of exercise on the epigenetic processes of this population.
Keywords
Autism spectrum disorder; DNA methylation; endurance training; exercise; reaction time.
Introducción
El trastorno de espectro autista (TEA) es un trastorno del neurodesarrollo que tiene fuertes bases genéticas, una etiología heterogénea y diversas presentaciones clínicas. Sus principales signos son el déficit en interacción social, en comunicación y los comportamientos o intereses repetitivos [1]. Así mismo, también se ha reportado bajo rendimiento en tareas motoras y déficit anticipatorio, que se explica principalmente por alteraciones a nivel cerebelar [2,3]. La anticipación resulta ser necesaria para el desarrollo de habilidades como la formación de modelos internos, la imitación e interpretación de acciones de otros y la generación de comportamientos durante la interacción social. Dichas respuestas anticipatorias se han visto reducidas en niños diagnosticados con TEA, lo que se ha relacionado con el comportamiento social característico de esta población [4].
El tiempo de reacción simple (TRS) puede definirse como el tiempo que pasa entre el estímulo y la reacción a este, y es mediado por diversos factores, como por ejemplo la velocidad de procesamiento. Además, el TRS se caracteriza por contar con un único estímulo y así mismo una única respuesta. Por ejemplo, aparece un objeto en la pantalla del computador y se presiona una tecla lo más rápido posible al aparecer dicho estímulo [5]. Dado lo anterior, el tiempo de TRS resulta una medida que puede dar cuenta de la capacidad de anticipación y además refleja el comportamiento motor, debido a las regularidades temporales en la ocurrencia de eventos de un estímulo. Dentro de la medición del TRS se pueden dar respuestas anticipatorias, en términos de milisegundos (ms) y segundos (s), definidas por debajo de los 100 ms y son respuestas planificadas e iniciadas antes de la aparición del objetivo [6]. Para niños con TEA se ha reportado previamente que el tiempo de reacción es significativamente más largo en diferentes tipos de tareas, incluyendo el TRS. Teniendo en cuenta la implicación funcional del TRS, puede ser que este enlentecimiento se relacione con las ya reconocidas alteraciones en el lenguaje y el desarrollo motor y social de los niños con TEA [7].
Por otro lado, el TEA presenta una alta heredabilidad, pero la tasa de concordancia entre gemelos monocigóticos puede variar entre 60-92% [8], sugiriendo que, junto con los mecanismos genéticos, los factores ambientales contribuyen a la aparición del trastorno. Lo anterior puede darse a través de mecanismos como la regulación epigenética, que se comprende como aquellos procesos moleculares que modulan la expresión genética sin cambios en la secuencia de ADN [9]. Uno de los mecanismos epigenéticos más estudiados es la metilación de ADN, que se refiere a la agregación de un grupo metilo a la posición C5 de la citosina base, y se da predominantemente en citosinas dentro de dinucleótidos CpG. Dicho proceso se da de forma importante durante el desarrollo en etapas de diferenciación y crecimiento de tejidos [10]. La metilación de ADN puede influir en la función y el fenotipo, aumentando o inhibiendo los niveles de expresión: un aumento en la metilación (hipermetilación) se relaciona con silenciamiento del gen; una disminución en la metilación (hipometilación), con aumento en la expresión del gen [11].
Respecto a lo anterior, el gen SHANK3 es uno de los genes más fuertemente relacionados con el TEA, gracias a estudios con modelos en ratón y también en estudios de vinculación familiar. SHANK3, conocido como ProSAP2, se localiza en la posición 22q13.3, tiene 22 exones y expresa 60kb de ADN. Es una proteína de andamiaje implicada en la densidad postsináptica (PSD), que forma un complejo en las sinapsis glutamatérgicas; este gen se expresa principalmente en corteza cerebral y cerebelo [12]. Además del uso de promotores alternativos y el empalme de ARNm, la expresión de SHANK3 está regulada por mecanismos epigenéticos como la metilación del ADN [13]. En tejidos cerebrales de personas con TEA, se ha reportado que SHANK3 tiene un patrón con tendencia a hipermetilación en sus islas 2 y 4, asociando la alteración en la metilación de ADN con la expresión del gen y la susceptibilidad al TEA [14].
Referente al ejercicio físico aeróbico (EFA), este se ha incluido como parte del tratamiento desde la fisioterapia para niños con TEA, encontrándose que influye en aspectos como habilidades sociales, comunicativas, motoras y cognitivas [15,16]. La evidencia sugiere que el tipo de ejercicio que favorece estos resultados es aquel de mayor intensidad [17]. De igual forma, se ha demostrado que el ejercicio físico puede tener efectos duraderos, a tal punto de influir en la regulación epigenética de diferentes genes tanto a nivel muscular como cerebral, y esto puede llegar a intervenir en el comportamiento y función a largo plazo [18]. Así mismo, algunos autores han descrito que gracias a la influencia del ejercicio en la epigenética (incluida la metilación de ADN) en el sistema nervioso central (SNC), es que se dan los beneficios a nivel cognitivo y comportamental [19].
Estudios en modelos animales han mostrado el potencial del EFA para alterar la metilación de ADN en sitios CpG en genes como el AMPa2 [20], en el factor neurotrófico derivado del cerebro (BDNF) y además en la expresión de genes [21]. También puede llegar a modificar el epigenoma del hipocampo de ratón [22]. De igual forma, en humanos se ha reportado que el ejercicio aeróbico genera cambios en los patrones de metilación y estos se asocian con reducción de la presión arterial [23], y así mismo existe evidencia sobre el efecto de este tipo de ejercicio en adultos mayores con deterioro cognitivo, en la metilación de genes como el VPS52, asociado al tráfico de proteínas intracelulares [24].
Finalmente, conociendo los efectos que se han descrito del EFA en términos sociales, motores y cognitivos para población con TEA, además de los potenciales efectos en la metilación de ADN, este estudio tuvo como objetivo analizar el efecto de un programa de EFA en el TRS y la metilación de ADN de la isla 2 del gen SHANK3 en niños con TEA.
Materiales y métodos
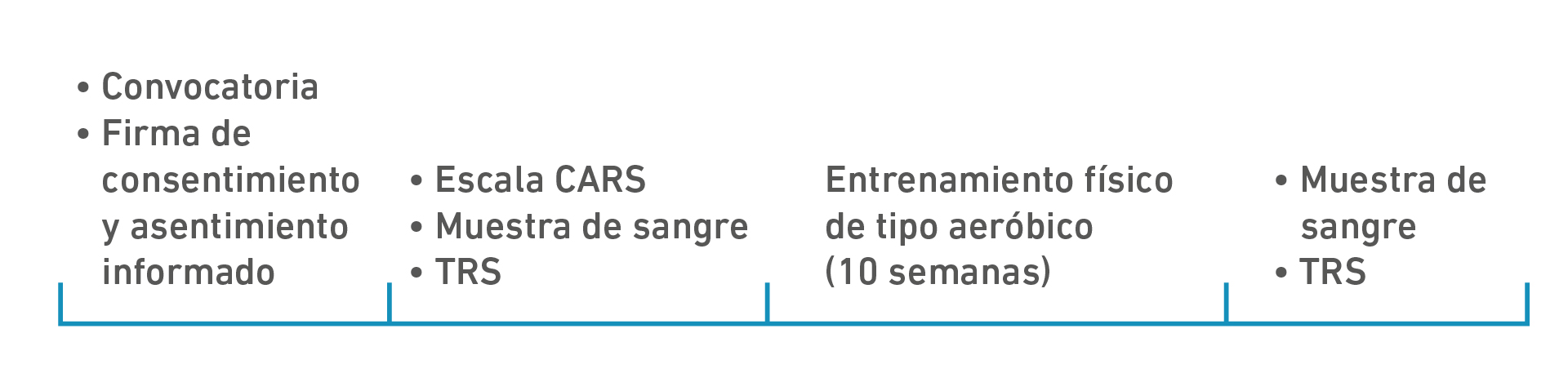
Se realizó un estudio de tipo cuasiexperimental para analizar el efecto de un programa de EFA en un grupo de niños diagnosticados con TEA. Los participantes pertenecían a una institución de la ciudad de Bogotá, especializada en el tratamiento de TEA. La convocatoria se realizó mediante charlas con cada uno de los acudientes y/o padres-madres de familia, aprobadas por los coordinadores de dicha institución, y una vez brindada la información se prosiguió con la firma del consentimiento informado. La participación de los niños fue voluntaria y se hizo uso de un asentimiento informado ajustado para las características de la población participante. Si alguno de los participantes manifestaba no querer participar en alguna de las actividades (extracción de sangre periférica o desarrollo de los ejercicios en más de una sesión), se retiraría del estudio. Inicialmente, se realizó una prueba de TRS y toma de muestra de sangre periférica. Se prosiguió con el entrenamiento durante 10 semanas, Finalmente, se volvió a evaluar el TRS y a extraer sangre periférica para los posteriores análisis (ver Figura 1).
Figura 1. Metodología del estudio.
Nota. Cuatro momentos del estudio. 1) Convocatoria y firma de documentos; 2) Aplicación de escala CARS, toma de muestra de sangre de periférica y evaluación del TR; 3) Aplicación protocolo de entrenamiento; 4) Segunda toma de muestra de sangre periférica y segunda evaluación del TRS, posentrenamiento. CARS: Childhood Autism Rating Scale; TRS: Tiempo de Reacción Simple.
Participantes
Niños con edades entre 7 y 11 años, con diagnóstico de TEA y con una puntuación de autismo “moderado” según la Childhood Autism Rating Scale (CARS) [25]. La escala fue aplicada después de la firma del consentimiento y el asentimiento informados, con apoyo de un profesional en psicología. Los criterios de inclusión fueron tener diagnóstico de TEA, tener entre 7 y 11 años, obtener una puntuación de 25-36 en la CARS, presentar lenguaje verbal, contar con la firma del consentimiento informado por parte de padres y/o tutores legales y haber firmado el asentimiento informado. Los criterios de no inclusión fueron presentar diagnósticos de síndromes cromosómicos o déficit cognitivo, presentar patologías que les impidieran realizar actividad física de alta intensidad, además de la manifestación verbal de no querer hacer parte de alguno de los procedimientos del estudio antes o durante el desarrollo de éste. La muestra inicial fue de 35 niños, de la cual se excluyeron 26 que no cumplían los criterios de edad, es decir, no presentaron comunicación verbal o firma del consentimiento informado por parte de padres y/o tutores legales. La muestra final fue de 9 participantes.
Protocolo de entrenamiento
El programa de EFA se realizó siguiendo las recomendaciones de la OMS [26], y la guía planteada por Srinivasan et al. en 2014 para desarrollar programas de entrenamiento para niños y niñas con TEA [27]. El protocolo usado en este estudio fue de 10 semanas, en las que se desarrollaron dos sesiones por semana, con una duración de 45 minutos cada una. Se llevaron a cabo actividades en circuitos motores, que incluyeron trotar, correr, saltar, rolar, entre otras. Para la prescripción de la carga se tuvo en cuenta la frecuencia cardiaca máxima (FCmáx), en la que los participantes debían alcanzar un 60-80% de esta, iniciando en 60% y progresando hasta alcanzar el 80%. La FCmáx fue calculada mediante la fórmula: 208 - (0.8 x edad) [28,29], teniendo en cuenta las características de la población. La frecuencia cardiaca (FC) se monitorizó mediante pulsooxímetría. No se usaron elementos como acelerómetros o relojes POLAR®, dado que la mayoría de los participantes no toleraban su uso (bandas y relojes) y los retiraban.
El diseño y aplicación del programa estuvo a cargo de una fisioterapeuta con experiencia en atención a población con TEA, y las actividades se llevaban a cabo de forma individual con cada niño. El programa se desarrolló en la misma institución donde los participantes recibían terapias de forma regular, ya que cuenta con un gimnasio amplio para realizar este tipo de actividades.
Análisis del tiempo de reacción simple (TRS)
Se midió el TRS antes y después del programa de entrenamiento con el software INQUISIT 4.0 [30,31], el cual se programó para que el estímulo (círculo rojo en el centro de la pantalla de fondo blanco) apareciera en diferentes lapsos de tiempo. Los participantes se ubicaron sentados frente a un computador con una pantalla de 14´´, a una distancia de 40 cm. Cada participante realizó 30 intentos (tanto en el momento pre como pos), y se tomó el mejor tiempo de los últimos 10 intentos en los dos momentos.
Análisis de metilación de ADN
Se realizó extracción de sangre periférica de los participantes, una semana antes y una semana después del programa de EFA, en el Instituto de Genética de la Universidad Nacional de Colombia, con apoyo de personal de enfermería capacitado para tal fin.
Se extrajo ADN genómico de la sangre periférica de los participantes con el kit ReliaPrep Blood gDNA Miniprep System ™ (A5082-PROMEGA), siguiendo el protocolo recomendado por el fabricante. Después se realizó la cuantificación del ADN en el espectrofotómetro Thermo Scientific NanoDrop 2000c, usando agua libre de nucleasas como blanco y finalmente se almacenó a -4°C [32].
Para determinar la metilación de los dinucleótidos CpG del ADN extraído, se sometió a una conversión con bisulfito de sodio, con el kit EZ DNA Methylation-Direct TM (Zymmo Research) y se almacenó a <-70°C para su uso posterior. Se utilizó la metodología Bisulphite Sequencing PCR (BSP) para evaluar la isla 2 del gen SHANK3, donde se analizaron 12 dinucleótidos CpG, mediante el software ESME (Epigenetic Sequencing Methyation analysis software) [32].
Los análisis de metilación de ADN fueron desarrollados en el Instituto de Genética de la Universidad Nacional de Colombia. En este mismo lugar se almacenaron las muestras, cuyo único uso fue para el presente estudio.
Análisis estadístico
Los datos fueron recolectados en una base de datos en Excel y se ingresaron al programa R. Inicialmente se usó una prueba ANOVA, y considerando las características de la población, finalmente se empleó la prueba no paramétrica Kruskall-Wallis para el análisis de los datos de metilación (porcentajes) y TRS pre y pos entrenamiento. Se usó un análisis de regresión lineal para evaluar en conjunto los resultados entre los porcentajes de metilación y el TRS. Se consideró un nivel de significancia p<0.05. Se calculó la mediana tanto para la edad como para el puntaje en la escala CARS.
Consideraciones éticas
La investigación fue aprobada por el Comité de Ética de la Facultad de Medicina de la Universidad Nacional de Colombia, mediante el acta No. 012-225-18. Para su realización se siguieron los principios éticos de investigación biomédica en seres humanos, de acuerdo con lo establecido en la Declaración de Helsinki, y así mismo se tuvo en cuenta lo estipulado en la Resolución No. 08430 de 1993 del Ministerio de salud de Colombia.
Resultados
Características de la población
La mediana de edad de los participantes fue de 9 años. El 100% de la población fue de sexo masculino y la mediana en la escala CARS fue de 30, lo que indica un nivel moderado de autismo en la población participante (Tabla 1).
Tabla 1. Caracterización de la población y tiempo de reacción simple antes y después del programa de entrenamiento
| Participantes | Edad | Sexo | CARS | TRS (ms) | TRS (ms) |
|---|---|---|---|---|---|
| PRE | POS | ||||
| 1 | 10 | M | 33.5 | 355 | 303 |
| 2 | 7 | M | 30 | 236 | 266 |
| 3 | 9 | M | 30.5 | 454 | 334 |
| 4 | 7 | M | 33.5 | 421 | 360 |
| 5 | 11 | M | 30.5 | 267 | 227 |
| 6 | 9 | M | 35.5 | 326 | 287 |
| 7 | 11 | M | 27.5 | 226 | 255 |
| 8 | 11 | M | 27 | 274 | 272 |
| 9 | 10 | M | 33 | 350 | 350 |
| Mediana | 9 | - | 30 | 326 | 287 |
| p Valor TRS | - | - | - | 0.53 | |
Nota. CARS (Childhood Autism Rating Scale); ms (milisegundos); p-valor del TRS antes y después. p<0.05. Fuente: Elaboración propia a partir de los datos obtenidos.
Tiempo de reacción simple
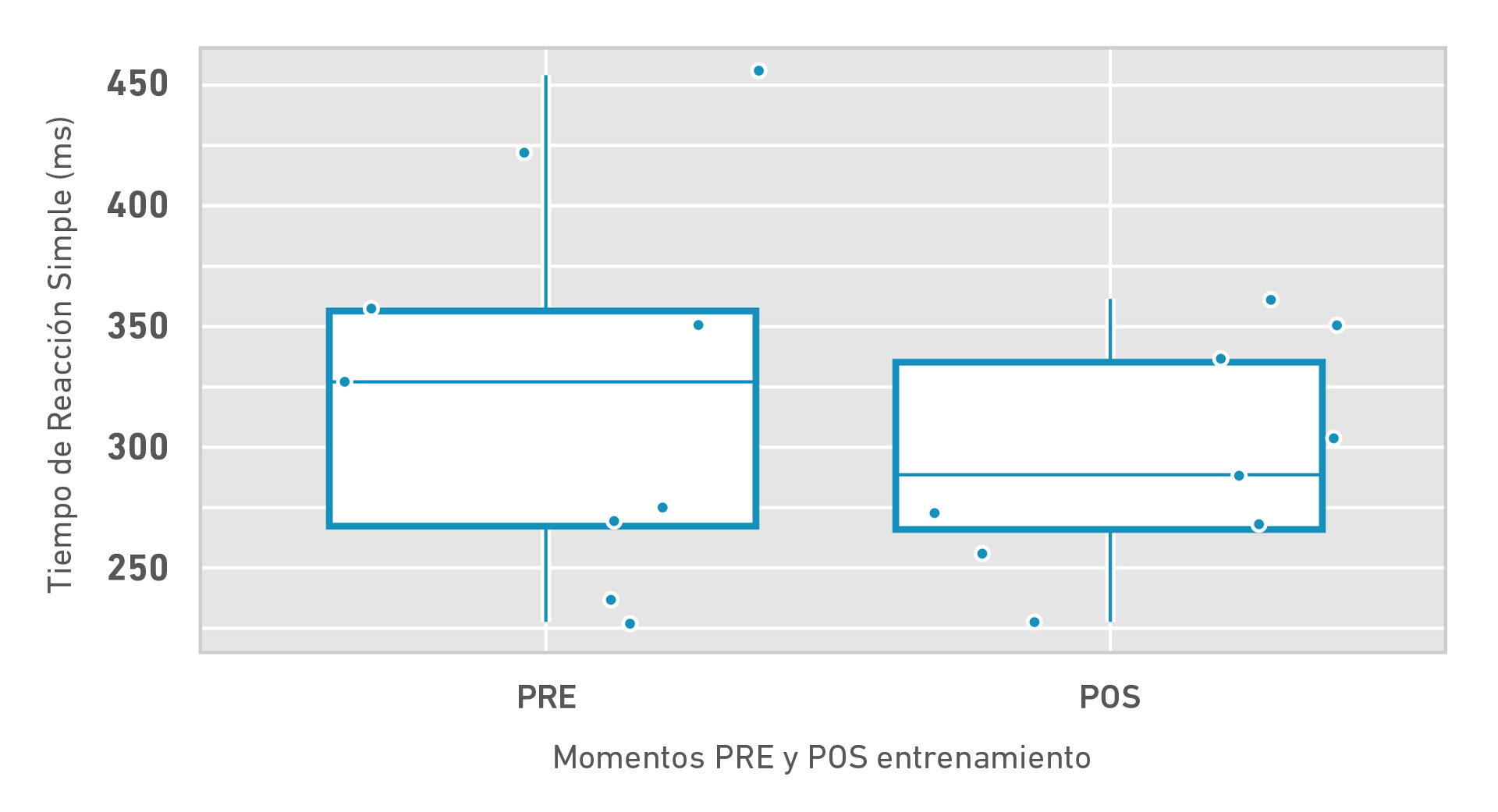
Mediante un Box-Plot (Figura 2), se evidencia que el TRS fue mayor antes del programa de entrenamiento y que hay una tendencia hacia su disminución en las mediciones posentrenamiento. Sin embargo, se encuentra que la dispersión es alta, lo que, combinado con una población tan pequeña, resulta en que el modelo de análisis de varianza no muestre diferencias significativas en el TRS (p=0.53) (ver Tabla 1).
Figura 2. Diagrama de dispersión del Tiempo de Reacción Simple PRE y POS entrenamiento.
Nota. Elaboración propia a partir de los datos obtenidos. ms: milisegundos
Uno de los participantes no presentó cambios y dos de ellos presentaron un TRS mayor después del programa de entrenamiento de EFA (Tabla 1).
Niveles de metilación en la isla 2 del gen SHANK3
Se encontró un patrón de hipermetilación en 11 de los dinucleótidos CpG analizados tanto en las muestras PRE como en las POS; uno de los dinucleótidos (CpG146) presentó un patrón de hipometilación. La posición CpG108 presentó una diferencia estadísticamente significativa en los resultados PRE y POS (Tabla 2).
Tabla 2. Resultados del análisis de metilación PRE y POS entrenamiento
| Región | Posición genómica de las CpG | Metilación (%) | p Valor (metilación PRE y POS) | P valor Regresión lineal (% metilación y TRS) | |
|---|---|---|---|---|---|
| PRE | POS | ||||
| 26 | ND | ND | ND | ND | |
| 59 | 100 | 100 | 0.725 | 0.219 | |
| 63 | 96 | 95 | 0.734 | 0.127 | |
| 73 | 100 | 100 | 0.880 | 0.147 | |
| 84 | 100 | 100 | 0.650 | 0.222 | |
| Isla 2- | 89 | 100 | 100 | 0.829 | 0.161 |
| Gen SHANK3 | 108 | 97 | 89 | 0.032* | 0.226 |
| chr22:51,135,671-51,136,118 | 123 | 94 | 91 | 0.523 | 0.090 |
| 128 | 94 | 96 | 0.244 | 0.079 | |
| 132 | 90 | 89 | 0.723 | 0.974 | |
| 146 | 68 | 47 | 0.093 | 0.992 | |
| 198 | 100 | 100 | 0.823 | 0.799 | |
| 207 | 93 | 93 | 0.391 | 0.289 | |
Nota. CpGs individuales, evaluadas mediante metodología BSP (Bisulphite Sequencing PCR) para la isla 2 del gen SHANK3; porcentajes de metilación (%) PRE y POS entrenamiento; p-Valor para diferencias de medias PRE y POS entrenamiento en cada CpG analizada. Chr22: Cromosoma 22. ND: No datos. *p-valor <0.05
Análisis entre el porcentaje de metilación y el TRS
Se uso un modelo de regresión lineal simple, en el que no se evidencia que alguna de las posiciones tenga relación estadísticamente significativa entre la diferencia del porcentaje de metilación y la diferencia en TRS (Tabla 1).
Discusión
En los últimos años se han descrito los diferentes efectos que tiene el ejercicio físico en el TEA, dentro de los cuales se encuentran los relacionados con mejoras en tareas cognitivas y motoras [33]. Asi mismo, se ha descrito el efecto del ejercicio en procesos epigenéticos [34,35], como la metilación de ADN, que podría ser uno de los factores que explican el efecto benéfico del ejercicio. Las investigaciones en esta materia se han realizado en diferentes patologías, como hipertensión arterial, diabetes y cáncer [36-38], pero no en TEA.
En el presente estudio se identificó una mediana de 326 ms y 287 ms para el TRS, en los momentos PRE y POS, respectivamente, lo cual indica una tendencia a la disminución. Los resultados del TRS por encima de 220 ms en este estudio son acordes con lo esperado para la población con TEA [39]. Sin embargo, aunque el TRS de la población en este estudio es bajo, en relación con otros resultados para niños con TEA, como por ejemplo el de Yair et al., en el que se reportan 369-357 ms para una tarea de TRS [40], sigue siendo alto, según lo esperado en niños de esas mismas edades (7-11 años), y sin alteraciones en el neurodesarrollo, que es un TRS de 200ms [41]. Este resultado, entonces, sería congruente con la alteración en la anticipación y disminución en TRS reportada para los niños con TEA [4].
Respecto a la tendencia en la disminución en el TRS después del programa de entrenamiento de 10 semanas implementado en la presente investigación, hay estudios reportados previamente que señalan una disminución en el TRS. Sin embargo, esta disminución ha sido reportada para población con TDAH y niños sin alteraciones en el neurodesarrollo. Por ejemplo, en el estudio de Ludyga et al., se encontró una diferencia de 58.9 ms en los TRS pre y posentrenamiento en niños de 12 a 15 años sin alteraciones en el neurodesarrollo [42]. Por su parte, Chuang et al. encontraron mejores TRS en niños con TDAH, después de una sesión de ejercicio de 30 min [43]. Dados los resultados en términos de TRS en el presente estudio, se puede inferir que una población de niños con TEA puede verse beneficiada con este tipo de entrenamiento y, en este sentido, sería importante correlacionar en futuras investigaciones el TRS con el desempeño en funciones ejecutivas, desarrollo social y movimientos estereotipados, teniendo en cuenta lo reportado por otros autores [15].
Por otra parte, mediante la metodología BSP, se logró identificar un patrón de hipermetilación en sangre periférica de pacientes pediátricos con TEA, en 11 de los 13 dinucleótidos de la isla 2 del gen SHANK3, con una mediana de 92% y 91% para la metilación pre y posentrenamiento, respectivamente. Este resultado coincide con lo reportado previamente en muestras de tejido cerebral, tanto para el gen SHANK3 [14] como para otros genes relacionados con el TEA, en los que ya se han encontrado dichos patrones de hipermetilación [44,45]. Este mismo patrón fue hallado en muestras de sangre periférica para SHANK3 en niños con TEA, en edades entre 2 a 13 años [46,47].
Los resultados del presente estudio indicarían que puede haber una baja expresión de la proteína codificada por el gen SHANK3, teniendo en cuenta que la hipermetilación se relaciona con el silenciamiento génico. En este sentido, para la población de este estudio en particular sería preciso señalar que la isla 2 del SHANK3 estaría involucrada como uno de los múltiples factores inmersos en la presentación del trastorno, teniendo en cuenta la relación reportada entre el gen SHANK3 y el TEA [48]. Al respecto, es necesario realizar estudios en los que, además del patrón de metilación, se analice la expresión del gen o genes candidatos, para poder correlacionar la metilación de ADN con la expresión génica.
Contrario a lo anterior, se encontró un patrón de metilación diferencial en la posición CpG146 en relación con las demás (68% pre y 47% pos, que correspondería a una hipometilación). Este resultado es acorde a lo reportado en la literatura, donde se han evidenciado diferencias en los niveles de metilación en sitios CpG de una misma isla y un mismo gen en gemelos monocigotos discordantes con TEA y en casos de TEA en comparación con controles [45,49]. En este sentido,el resultado de este estudio podría aportar información al conocimiento sobre la implicación de diferentes posiciones CpG en el funcionamiento del SHANK3 y su correlación con la etiología del TEA.
En relación con el efecto del programa de EFA en la metilación de ADN, se encontró una diferencia estadísticamente significativa en los resultados pre y posentrenamiento de la posición CpG108 (p = 0.032). Respecto a esto, autores como Rodrigues et al. describen en modelos animales el potencial del ejercicio como modulador de la metilación de ADN ante condiciones de estrés, encontrando menores niveles de metilación después de un entrenamiento físico [50]. El hallazgo en el presente estudio resulta importante, teniendo en cuenta la cantidad de participantes y el tiempo de duración del programa de entrenamiento. Sin embargo, sería conveniente que en un futuro se evalúe más a profundidad esta posición y las posibles implicaciones funcionales para la proteína expresada por SHANK3 en niños y niñas con TEA. Así mismo, los resultados encontrados dan cuenta del efecto del EFA en los niveles de metilación para la población estudiada, y se podría deducir que este sería específico para ciertas posiciones dentro de las islas CpG y no para la isla en su totalidad.
Ahora bien, se ha descrito que SHANK3 se expresa en diferentes zonas del cerebro [12], y es posible que su expresión (en conjunto con la expresión de otros genes) se relacione con el funcionamiento cerebral [48], lo cual explicaría las alteraciones comportamentales y sociales en población con TEA. En ese sentido, promover cambios en la metilación del gen podría, a su vez, generar un cambio en respuestas cognitivas, motoras, y sociales, que se evidenciarían en tareas como la del TRS.
En este estudio se buscó analizar los efectos en la metilación de la isla 2 del gen SHANK3 a partir de un entrenamiento aeróbico y su correlación con el TRS en niños con TEA. Dado que no se encontró una relación estadísticamente significativa entre las variables, es posible decir que la isla 2 del gen SHANK3 no estaría relacionada con la anticipación medida a través del TRS en esta población específica, lo cual tendría sentido, dado que el TEA es un trastorno multifactorial y posiblemente el gen SHANK3 no sea el único implicado en su desarrollo.
Respecto a la relación entre la metilación de ADN y el ejercicio, hay estudios realizados en humanos que describen cambios tanto hacia la hipometilación como hacia la hipermetilación en genes de regulación celular asociados con la realización de actividad física [51]. De igual forma, hay estudios que reportan una correlación entre la metilación y el nivel de actividad física, en personas que presentan patologías como cáncer de seno [52]. No obstante, debe mencionarse que dichos estudios, además de haberse realizado en una población diferente a la del presente estudio, analizan otros genes, y algunos de ellos están relacionados con regulación celular o con otros tejidos como el muscular.
Finalmente, cabe resaltar que a partir de la similitud del patrón de metilación del presente estudio (muestras de sangre periférica) y lo reportado en tejido cerebral, se podría pensar en la sangre como un posible medio para conocer parcialmente lo que pasa en el cerebro a nivel epigenético, pues varios autores han descrito una correlación entre la metilación en el cerebro y la sangre periférica [53,54].
Limitaciones y fortalezas
Este estudio presenta limitaciones en términos del tamaño de la muestra y de los genes estudiados, dado que hay varios genes que pueden estar involucrados en el desarrollo del TEA. Sin embargo, se resalta la novedad en términos del análisis de los posibles cambios de metilación de ADN en niños con TEA y su relación con el TRS después de un entrenamiento de tipo aeróbico. Cabe destacar también que este es el primer estudio en Colombia donde se implementa un programa de entrenamiento físico estructurado para población pediátrica con diagnóstico de TEA y se evalúa conjuntamente el TRS y el comportamiento de la metilación de ADN en un gen específico.
Conclusiones
El TRS en niños con TEA puede verse modificado con tendencia a la disminución, gracias a un entrenamiento de tipo aeróbico durante 10 semanas. Este resultado puede ser la base para implementar este tipo de programas en población con TEA y evaluar si a largo plazo podría beneficiar no solo la anticipación, sino su relación con el desarrollo social y cognitivo.
Un programa de entrenamiento basado en EFA, de intensidad moderada a vigorosa, tiene un efecto únicamente en la posición CpG108, y en ese sentido, para esta población específica, la isla 2 del gen SHANK3 no es susceptible al ejercicio.
Para este grupo de niños en particular, la metilación en la isla 2 del gen SHANK3 no estaría relacionada con la respuesta anticipatoria medida en términos de TRS, antes y después de un entrenamiento físico.
El desarrollo de este tipo de investigaciones resulta importante para fortalecer el posicionamiento de la fisioterapia en el tratamiento del TEA y es una posibilidad de fundamentar desde las ciencias básicas los diferentes métodos y técnicas de intervención, ya que aún no se conocen los mecanismos exactos por los cuales se dan los beneficios relacionados con el ejercicio físico.
Los resultados presentados en este estudio no deben ser tomados a la ligera y es necesario realizar futuras investigaciones con un tamaño poblacional más grande, en las que se analice cómo el ejercicio puede influir en el comportamiento de la metilación de las otras islas del gen SHANK3, así como otros genes relacionados con el TEA y diferentes variables (función cognitiva, desarrollo social y aspectos comportamentales).
Referencias
1. Li D, Karnath HO, Xu X. Candidate Biomarkers in Children with Autism Spectrum Disorder: A Review of MRI Studies. Neurosci Bull [Internet]. 2017 [citado 2022 nov 6];33(2):219-37. doi: https://doi.org/10.1007/s12264-017-0118-1
2. Mosconi XMW, Mohanty S, Greene RK, Cook EH, Vaillancourt DE, Sweeney JA. Feedforward and Feedback Motor Control Abnormalities Implicate Cerebellar Dysfunctions in Autism Spectrum Disorder. The Journal of neuroscience [Internet]. 2015 [citado 2022 oct 31];35(5):2015-25. doi: https://doi.org/10.1523/JNEUROSCI.2731-14.2015
3. Gidley Larson JC, Bastian AJ, Donchin O, Shadmehr R, Mostofsky SH. Acquisition of internal models of motor tasks in children with autism. Brain [Internet]. 2008 [citado 2022 nov 6];131(11):2894-903. doi: https://doi.org/10.1093/brain/awn226
4. Feller C, Dubois C, Eliez S, Schneider M. Episodic Future Thinking in Autism Spectrum Disorder and 22q11.2 Deletion Syndrome: Association with Anticipatory Pleasure and Social Functioning. J Autism Dev Disord [Internet]. 2021 dic 1 [citado 2022 nov 5];51(12):4587-604. doi: https://doi.org/10.1007/s10803-021-04903-2
5. Zou T, Cao S, Liu W, Li L, Jiang J, Wu L. Is simple reaction time or choice reaction time an indicator of all-cause mortality or CVD mortality? [Internet]. Public Health. 2021 [citado 2022 nov 5];199:34-41. doi: https://doi.org/10.1016/j.puhe.2021.07.045
6. Debrabant J, Gheysen F, Vingerhoets G, van Waelvelde H. Age-related differences in predictive response timing in children: Evidence from regularly relative to irregularly paced reaction time performance. Hum Mov Sci [Internet]. 2012 [citado 2022 nov 6];31(4):801-10. doi: https://doi.org/10.1016/j.humov.2011.09.006
7. Zapparrata NM, Brooks PJ, Ober TM. Slower Processing Speed in Autism Spectrum Disorder: A Meta-analytic Investigation of Time-Based Tasks. J Autism Dev Disord [Internet]. 2022 [citado 2022 nov 6]. doi: https://doi.org/10.1007/s10803-022-05736-3
8. Lopes Cardoso I, Almeida S. Genes Involved in the Development of autism. Int Arch Commun Disord [Internet]. 2019 [citado 2022 nov 6];2:11. doi: https://doi.org/10.23937/IACOD-2017/1710011
9. Halabian R, Valizadeh Arshad, Ahmadi A, Saeedi P, Azimzadeh Jamalkandi S, Alivand MR. Laboratory methods to decipher epigenetic signatures: a comparative review [Internet]. Cellular and Molecular Biology Letters. 2021 [citado 2022 nov 6];26:Article42. doi: https://doi.org/10.1186/s11658-021-00290-9
10. Mouat JS, LaSalle JM. The Promise of DNA Methylation in Understanding Multigenerational Factors in Autism Spectrum Disorders [Internet]. Frontiers in Genetics. 2022 [citado 2022 nov 6];13. doi: https://doi.org/10.3389/fgene.2022.831221
11. Mastroeni D, Grover A, Delvaux E, Whiteside C, Coleman PD, Rogers J. Epigenetic mechanisms in Alzheimer’s disease. Neurobiol Aging [Internet]. 2011 [citado 2022 nov 6];32(7):1161-80. doi: https://doi.org/10.1016/j.neurobiolaging.2010.08.017
12. Monteiro P, Feng G. SHANK proteins: roles at the synapse and in autism spectrum disorder. Nat Rev Neurosci [Internet]. 2017;18(3):147-57. doi: https://doi.org/10.1038/nrn.2016.183
13. Srancikova A, Reichova A, Bacova Z, Bakos J. Gene expression levels of DNA methyltransferase enzymes in Shank3-deficient mouse model of autism during early development. Endocr Regul [Internet]. 2021 oct 1 [citado 2022 nov 6];55(4):234-7. doi: https://doi.org/10.2478/enr-2021-0025
14. Zhu L, Wang X, Li XL, Towers A, Cao X, Wang P, et al. Epigenetic dysregulation of SHANK3 in brain tissues from individuals with autism spectrum disorders. Hum Mol Genet [Internet]. 2014 [citado 2022 nov 6];23(6):1563-78. doi: https://doi.org/10.1093/hmg/ddt547
15. Huang J, Du C, Liu J, Tan G. Meta-Analysis on Intervention Effects of Physical Activities on Children and Adolescents with Autism. Int J Environ Res Public Health [Internet]. 2020 mar 17 [citado 2022 nov 6];17(6). doi: https://doi.org/10.3390/ijerph17061950
16. Liang X, Li R, Wong SHS, Sum RKW, Wang P, Yang B, et al. The Effects of Exercise Interventions on Executive Functions in Children and Adolescents with Autism Spectrum Disorder: A Systematic Review and Meta-analysis [Internet]. Sports Medicine. 2022 [citado 2022 nov 6];52:75-88. doi: https://doi.org/10.1007/s40279-021-01545-3
17. Tse A. Brief Report: Impact of a Physical Exercise Intervention on Emotion Regulation and Behavioral Functioning in Children with Autism Spectrum Disorder. J Autism Dev Disord [Internet]. 2020 nov 1 [citado 2022 nov 6];50(11):4191-8. doi: https://doi.org/10.1007/s10803-020-04418-2
18. Haupt S, Niedrist T, Sourij H, Schwarzinger S, Moser O. The Impact of Exercise on Telomere Length, DNA Methylation and Metabolic Footprints [Internet]. Cells. 2022 [citado 2022 nov 6];11(1):153. doi: https://doi.org/10.3390/cells11010153
19. Liang J, Wang H, Zeng Y, Qu Y, Liu Q, Zhao F, et al. Physical exercise promotes brain remodeling by regulating epigenetics, neuroplasticity and neurotrophins. Rev Neurosci [Internet]. 2021 ago 1 [citado 2022 nov 6];32(6):615-29. doi: https://doi.org/10.1515/revneuro-2020-0099
20. King-Himmelreich TS, Schramm S, Wolters MC, Schmetzer J, Möser C v., Knothe C, et al. The impact of endurance exercise on global and AMPK gene-specific DNA methylation. Biochem Biophys Res Commun [Internet]. 2016 [citado 2022 nov 6];474(2):284-90. doi: https://doi.org/10.1016/j.bbrc.2016.04.078
21. Tomiga Y, Sakai K, Ra S-G, Kusano M, Ito A, Uehara Y, et al. Short-term running exercise alters DNA methylation patterns in neuronal nitric oxide synthase and brain-derived neurotrophic factor genes in the mouse hippocampus and reduces anxiety-like behaviors. Federation of American Societies for Experimental Biology [Internet]. 2021 abr [citado 2022 nov 6];35(8):1-45. doi: https://doi.org/10.1096/fj.202100630R
22. Urdinguio RG, Tejedor JR, Fernández-Sanjurjo M, Pérez RF, Peñarroya A, Ferrero C, et al. Physical exercise shapes the mouse brain epigenome. Mol Metab [Internet]. 2021 dic 1 [citado 2022 nov 6];54. doi: https://doi.org/10.1016/j.molmet.2021.101398
23. Ferrari L, Vicenzi M, Tarantini L, Barretta F, Sironi S, Baccarelli AA, et al. Effects of physical exercise on endothelial function and DNA methylation. Int J Environ Res Public Health [Internet]. 2019 [citado 2022 nov 9];16(14):Article2530. doi: https://doi.org/10.3390/ijerph16142530
24. Ngwa JS, Nwulia E, Ntekim O, Bedada FB, Kwabi-Addo B, Nadarajah S, et al. Aerobic Exercise Training-Induced Changes on DNA Methylation in Mild Cognitively Impaired Elderly African Americans: Gene, Exercise, and Memory Study - GEMS-I. Front Mol Neurosci [Internet]. 2022 ene 17 [citado 2022 nov 9];14. doi: https://doi.org/10.3389/fnmol.2021.752403
25. Breidbord J, Croudace TJ. Reliability generalization for childhood autism rating scale. J Autism Dev Disord [Internet]. 2013 [citado 2022 nov 6];43(12):2855-65. doi: https://doi.org/10.1007/s10803-013-1832-9
26. Bull FC, Al-Ansari SS, Biddle S, Borodulin K, Buman MP, Cardon G, et al. World Health Organization 2020 guidelines on physical activity and sedentary behaviour [Internet]. British Journal of Sports Medicine. 2020 [citado 2022 oct 5];54:1451-62. doi: https://doi.org/10.1136/bjsports-2020-102955
27. Srinivasan SM, Pescatello LS, Bhat AN. Current Perspectives on Physical Activity and Exercise Recommendations for Children and Adolescents With Autism Spectrum Disorders. Phys Ther [Internet]. 2014 [citado 2022 oct 31];94(6):875-89. doi: https://doi.org/10.2522/ptj.20130157
28. Marjerrison AD, Woodruff ME, Hanna LE. Evaluating the prediction of maximal heart rate in children and adolescents. Res Q Exerc Sport [Internet]. 2010 [citado 2022 nov 4];81(4):466-71. doi: https://doi.org/10.1080/02701367.2010.10599707
29. Machado FA, Denadai BS. Validity of Maximum Heart Rate Prediction Equations for Children and Adolescents. Arq Bras Cardiol [Internet]. 2011 [citado 2022 oct 29];97(2):136-40. doi: https://doi.org/10.1590/S0066-782X2011005000078
30. Liverani MC, Manuel AL, Nahum L, Guardabassi V, Tomasetto C, Schnider A. Children’s sense of reality: The development of orbitofrontal reality filtering. Child Neuropsychology [Internet]. 2017 may 19 [citado 2022 nov 4];23(4):408-21. doi: https://doi.org/10.1080/09297049.2015.1120861
31. Dave J, Grover P, Deo M. Submaximal exercises cause immediate reduction in the visual reaction time in normal individuals. Journal of Society of Indian Physiotherapists [Internet]. 2021 [citado 2022 nov 1];5(2):59. doi: https://doi.org/10.4103/jsip.jsip_8_21
32. Salcedo-Tacuma D, Melgarejo JD, Mahecha MF, Ortega-Rojas J, Carlos †, Arboleda-Bustos E, et al. Differential Methylation Levels in CpGs of the BIN1 Gene in Individuals With Alzheimer Disease [Internet]. Alzheimer Disease & Associated Disorders. 2019;33(4):p 321-326. doi: https://doi.org/10.1097/WAD.0000000000000329
33. Pan CY, Chu CH, Tsai CL, Sung MC, Huang CY, Ma WY. The impacts of physical activity intervention on physical and cognitive outcomes in children with autism spectrum disorder. Autism [Internet]. 2017 [citado 2022 nov 6];21(2):190-202. doi: https://doi.org/10.1177/1362361316633562
34. Fernandes J, Arida RM, Gomez-Pinilla F. Physical exercise as an epigenetic modulator of brain plasticity and cognition. Neurosci Biobehav Rev [Internet]. 2017 sep [citado 2021 ago 12];80:443-56. doi: https://doi.org/10.1016/j.neubiorev.2017.06.012
35. Renó UP, Soares SF, Penteado JL, Casanova A, Nóbrega C, de Oliveira EM. Exercise training and epigenetic regulation: Multilevel modification and regulation of gene expression. En: Wim E. Crusio, Haidong Dong, Heinfried H, editores. Advances in Experimental Medicine and Biology [Internet]. Springer Singapore; 2017 [citado 2022 nov 6]. p. 281-322. doi: https://doi.org/10.1007/978-981-10-4304-8_16
36. Wu G, Zhang X, Gao F. The epigenetic landscape of exercise in cardiac health and disease [Internet]. Journal of Sport and Health Science. 2021 [citado 2022 nov 9];10:648-59. doi: https://doi.org/10.1016/j.jshs.2020.12.003
37. Plaza-Diaz J, Izquierdo D, Torres-Martos Á, Baig AT, Aguilera CM, Ruiz-Ojeda FJ. Impact of Physical Activity and Exercise on the Epigenome in Skeletal Muscle and Effects on Systemic Metabolism. Biomedicines [Internet]. 2022 ene 1 [citado 2022 nov 9];10(1). doi: https://doi.org/10.3390/biomedicines10010126
38. Wagner MA, Erickson KI, Bender CM, Conley YP. The Influence of Physical Activity and Epigenomics On Cognitive Function and Brain Health in Breast Cancer [Internet]. Frontiers in Aging Neuroscience. 2020 [citado 2022 nov 9];12. doi: https://doi.org/10.3389/fnagi.2020.00123
39. Brodeur DA, Stewart J, Dawkins T, Burack JA. Utilitarian Attention by Children with Autism Spectrum Disorder on a Filtering Task. J Autism Dev Disord [Internet]. 2018 [citado 2022 ago 6];48:4019-4027. doi: https://doi.org/10.1007/s10803-018-3619-5
40. Bar-haim Y, Shulman C, Lamy D, Reuveni A. Attention to Eyes and Mouth in High-Functioning Children with Autism. 2006 [citado 2022 ago 6];36(1). doi: https://doi.org/10.1007/s10803-005-0046-1
41. Bucsuházy K, Semela M. Case Study: Reaction Time of Children According to Age. Procedia Eng [Internet]. 2017 [citado 2022 ago 6];187:408-13. doi: https://doi.org/10.1016/j.proeng.2017.04.393
42. Ludyga S, Gerber M, Herrmann C, Brand S, Pühse U. Chronic effects of exercise implemented during school-break time on neurophysiological indices of inhibitory control in adolescents. Trends Neurosci Educ [Internet]. 2018 [citado 2022 sep 6];10(June 2017):1-7. doi: https://doi.org/10.1016/j.tine.2017.11.001
43. Chuang LY, Tsai YJ, Chang YK, Huang CJ, Hung TM. Effects of acute aerobic exercise on response preparation in a Go/No Go Task in children with ADHD: An ERP study. J Sport Health Sci [Internet]. 2015 [citado 2022 ago 6];4(1):82-8. doi: https://doi.org/10.1016/j.jshs.2014.11.002
44. James SJ, Shpyleva S, Melnyk S, Pavliv O, Pogribny IP. Elevated 5-hydroxymethylcytosine in the Engrailed-2 (EN-2) promoter is associated with increased gene expression and decreased MeCP2 binding in autism cerebellum. Transl Psychiatry [Internet]. 2014 [citado 2022 ago 6];4(9):e460. doi: https://doi.org/10.1038/tp.2014.87
45. Wong CCY, Meaburn EL, Ronald A, Price TS, Jeffries AR, Schalkwyk LC, et al. Methylomic analysis of monozygotic twins discordant for autism spectrum disorder and related behavioural traits. Mol Psychiatry [Internet]. 2014 [citado 2022 ago 6];19(4):495-503. doi: https://doi.org/10.1038/mp.2013.41
46. Ching TT, Maunakea AK, Jun P, Hong C, Zardo G, Pinkel D, et al. Epigenome analyses using BAC microarrays identify evolutionary conservation of tissue-specific methylation of SHANK3. Nat Genet [Internet]. 2005 [citado 2022 ago 6];37(6):645-51. doi: https://doi.org/10.1038/ng1563
47. Tsang SY, Ahmad T, Mat FWK, Zhao C, Xiao S, Xia K, et al. Variation of global DNA methylation levels with age and in autistic children. Hum Genomics [Internet]. 2016 [citado 2022 ago 6];10(1):31. doi: https://doi.org/10.1186/s40246-016-0086-y
48. Bruno LP, Doddato G, Valentino F, Baldassarri M, Tita R, Fallerini C, et al. New candidates for autism/intellectual disability identified by whole-exome sequencing. Int J Mol Sci [Internet]. 2021 dic 1 [citado 2022 nov 6];22(24). doi: https://doi.org/10.3390/ijms222413439
49. Andrews S v., Sheppard B, Windham GC, Schieve LA, Schendel DE, Croen LA, et al. Case-control meta-analysis of blood DNA methylation and autism spectrum disorder. Mol Autism [Internet]. 2018 [citado 2022 ago 6];9(1):1-11. doi: https://doi.org/10.1186/s13229-018-0224-6
50. Rodrigues GM, Toffoli L v., Manfredo MH, Francis-Oliveira J, Silva AS, Raquel HA, et al. Acute stress affects the global DNA methylation profile in rat brain: Modulation by physical exercise. Behavioural Brain Research [Internet]. 2015 [citado 2022 ago 6];279:123-8. doi: https://doi.org/10.1016/j.bbr.2014.11.023
51. Światowy WJ, Drzewiecka H, Kliber M, Sąsiadek M, Karpiński P, Pławski A, et al. Physical activity and DNA methylation in humans [Internet]. International Journal of Molecular Sciences. 2021 [citado 2022 nov 6];22(23):Article12989. doi: https://doi.org/10.3390/ijms222312989
52. Gillman AS, Helmuth T, Koljack CE, Hutchison KE, Kohrt WM, Bryan AD. The effects of exercise duration and intensity on breast cancer-related dna methylation: A randomized controlled trial. Cancers (Basel) [Internet]. 2021 ago 2 [citado 2022 nov 6];13(16). doi: https://doi.org/10.3390/cancers13164128
53. Stevenson AJ, McCartney DL, Gadd DA, Shireby G, Hillary RF, King D, et al. A comparison of blood and brain-derived ageing and inflammation-related DNA methylation signatures and their association with microglial burdens. European Journal of Neuroscience [Internet]. 2022 [citado 2022 nov 6]. doi: https://doi.org/10.1111/ejn.15661
54. Braun PR, Han S, Hing B, Nagahama Y, Gaul LN, Heinzman JT, et al. Genome-wide DNA methylation comparison between live human brain and peripheral tissues within individuals. Transl Psychiatry [Internet]. 2019 ene 31 [citado 2022 ago 6];9(47). doi: https://doi.org/10.1038/s41398-019-0376-y